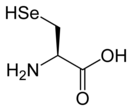
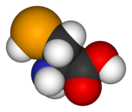
Селеноцистеин
| Селеноцистеин | |||
|---|---|---|---|
| |||
| Общие | |||
| Сокращения | Sec | ||
| Хим. формула | C3H7NO2Se | ||
| Физические свойства | |||
| Молярная масса | 168.053 г/моль г/моль | ||
| Классификация | |||
| Рег. номер CAS | 10236-58-5 | ||
| PubChem | 25076 | ||
| Рег. номер EINECS | 808-428-7 | ||
| SMILES | |||
| InChI | |||
| ChEBI | 16633 | ||
| ChemSpider | 23436 | ||
| Приведены данные для стандартных условий (25 °C, 100 кПа), если не указано иное. | |||
Селеноцистеи́н (сокращённо Sec или U, в старых публикациях также Se-Cys[1]) — 21-я протеиногенная аминокислота, аналог цистеина с заменой атома серы на атом селена (то есть серосодержащая тиольная группа заменена на селеносодержащую селенольную группу). Входит в состав активного центра фермента глутатионпероксидазы, а также в состав селенопротеинов[2], деиодаз и некоторых других белков. На мРНК селеноцистеин кодируется терминирующим кодоном UGA при условии, что за ним следует особая стимулирующая последовательность нуклеотидов.
Селеноцистеин впервые был обнаружен в бактериях клостридии в 1972 году биохимиком Треcой Штадтман (англ. Thressa Stadtman) из Национального института сердца, легких и крови США (Национальные институты здравоохранения США)[3]. Позже она с коллегами показала важную роль селеноцистеина в формировании многих других ферментов и его участие в метаболизме человека.
Структура[править | править код]
Структура селеноцистеина сходна с таковой цистеина с тем лишь отличием, что в нём атом серы заменён на атом селена, образуя селенольную группу, депротонированную при физиологических значениях pH. Белки, содержащие один или более остатков селеноцистеина, называются селенопротеинами. Они обладают каталитической активностью, обусловленной биохимической активностью селеноцистеина, из-за чего называются селеноэнзимами. У селеноэнзимов, структура которых была описана, были обнаружены триплеты аминокислот с каталитической активностью, которые обусловливают нуклеофильность активного сайта селеноцистеина.
Биология[править | править код]

Селеноцистеин имеет более низкую, чем у цистеина, константу диссоциации (5,47) и более высокий восстановительный потенциал. Благодаря этим свойствам селеноцистеин задействован в белках, обладающих антиоксидантной активностью[4].
В отличие от других аминокислот, встречающихся в белках, селеноцистеин не имеет своего особого кодона в генетическом коде[5]. В действительности он особым образом кодируется кодоном UGA, который обычно является стоп-кодоном. Такой механизм называется трансляционным перекодированием[6], а его эффективность зависит от синтезируемого селенопротеина и факторов инициации трансляции[7]. Если клетки живут в условиях отсутствия селена, то трансляция селенопротеина завершается на кодоне UGA, что приводит к образованию «обрезанного», нефункционального фермента. Кодон UGA кодирует селеноцистеин, если в мРНК присутствует последовательность вставки селеноцистеина (англ. SECIS element, SECIS). Элемент SECIS можно определить по характерным нуклеотидным последовательностям и особенностям вторичной структуры мРНК в области этого элемента. У бактерий элемент SECIS располагается непосредственно за кодоном UGA (в одной с ним рамке считывания)[8]. У архей и эукариот SECIS располагается в 3'-нетранслируемой области (англ. 3' untranslated region, 3' UTR) и может заставлять несколько кодонов UGA кодировать селеноцистеин[9].
Другое отличие селеноцистеина от стандартных аминокислот заключается в том, что он не существует в свободном виде внутри клетки, так как его высокая реакционная активность может нанести вред клетке. Вместо этого клетка хранит селен в форме менее активного селенида (H2Se). Синтез селеноцистеина осуществляется на специализированных тРНК, которые также включают его в нарастающую пептидную цепь. Первичная и вторичная структура селеноцистеин-специфичных тРНК, тРНКSec, отличаются от таковых у стандартных тРНК в нескольких аспектах. Так, акцепторная область содержит 8 пар оснований у бактерий и 10 — у эукариот, более длинную Т-петлю; помимо этого, для тРНКSec характерна замена нескольких довольно консервативных пар оснований. тРНКSec изначально связывается с серином при помощи фермента серил-тРНК лигазы, однако образующийся комплекс Ser-тРНКSec не вступает в трансляцию, поскольку не распознается нормальными трансляционными факторами (EF-Tu у бактерий и eEF1A у эукариот). Остаток серина, связанный тРНК, превращается в остаток селеноцистеина пиридоксальсодержащим ферментом селеноцистеинсинтазой. Наконец, образовавшийся комплекс Sec-тРНКSec специфично связывается с альтернативным трансляционным фактором (SelB или mSelB (или eEFSec)), который целенаправленно доставляет его в рибосому, транслирующую мРНК для селенопротеина. Специфичность этой доставки обусловлена наличием дополнительного белкового домена (у бактерий, SelB) или дополнительной субъединицы (SBP2 для эукариотической mSelB/eEFSec), которая связывается с соответствующим элементом вторичной структуры мРНК, образуемым элементом SECIS.
У человека известно 25 селенопротеинов[10].
Производные селеноцистеина γ-глутамил-Se-метилселеноцистеин и Se-метилселеноцистеин в природе известны у растений родов лук (Allium) и капуста (Brassica)[11].
Применение[править | править код]
Биотехнологическое применение селеноцистеина включает использование Sec, меченного изотопом 73Se (период полураспада 7,2 часа) в позитронно-эмиссионной томографии, а также Sec, содержащего 75Se (период полураспада 118,5 дней), для радиоактивного мечения. Один селеноцистеин или селеноцистеин в сочетании с селенометионином (SeMet) для облегчения фазы детерминации при помощи мультиволновой аномальной дисперсии в рентгеноструктурном анализе белков. Возможно включение стабильного изотопа 77Se, чей ядерный спин равен ½, для ядерного магнитного резонанса с высокой разрешающей способностью[2].
См. также[править | править код]
- Пирролизин, другая нестандартная аминокислота.
- Селенометионин, другая селеносодержащая аминокислота, случайно заменяющая метионин.
- Селенопротеины
Примечания[править | править код]
- ↑ IUPAC-IUBMB Joint Commission on Biochemical Nomenclature (JCBN) and Nomenclature Committee of IUBMB (NC-IUBMB) (англ.) // European Journal of Biochemistry : journal. — 1999. — Vol. 264, no. 2. — P. 607—609. — doi:10.1046/j.1432-1327.1999.news99.x. Архивировано 19 февраля 2018 года.
- ↑ 1 2 Johansson, L.; Gafvelin, G.; Amér, E. S. J. Selenocysteine in Proteins — Properties and Biotechnological Use (нем.) // Biochimica et Biophysica Acta : magazin. — 2005. — Bd. 1726, Nr. 1. — S. 1—13. — doi:10.1016/j.bbagen.2005.05.010.
- ↑ Stadtman T. Selenium biochemistry. — 1974. — Vol. 183, № 4128. — P. 915—922. — doi:10.1126/science.183.4128.915.
- ↑ Byun, B. J.; Kang, Y. K. Conformational Preferences and pKa Value of Selenocysteine Residue (англ.) // Biopolymer : journal. — 2011. — Vol. 95, no. 5. — P. 345—353. — doi:10.1002/bip.21581. — PMID 21213257.
- ↑ Böck A.; Forchhammer, K.; Heider, J.; Baron, C. Selenoprotein Synthesis: An Expansion of the Genetic Code (англ.) // Trends in Biochemical Sciences : journal. — Cell Press, 1991. — Vol. 16, no. 12. — P. 463—467. — doi:10.1016/0968-0004(91)90180-4. — PMID 1838215.
- ↑ Baranov P. V.; Gesteland R. F.; Atkins, J. F. Recoding: Translational Bifurcations in Gene Expression (англ.) // Gene : journal. — Elsevier, 2002. — Vol. 286, no. 5. — P. 187—201. — doi:10.1016/S0378-1119(02)00423-7. — PMID 11943474.
- ↑ Donovan, J.; Copeland, P. R. The Efficiency of Selenocysteine Incorporation is Regulated by Translation Initiation Factors (англ.) // Journal of Molecular Biology : journal. — 2010. — Vol. 400, no. 4. — P. 659—664. — doi:10.1016/j.jmb.2010.05.026. — PMID 20488192.
- ↑ Atkins, J. F. Recoding: Expansion of Decoding Rules Enriches Gene Expression (англ.). — Springer, 2009. — P. 31. — ISBN 9780387893815. Архивировано 6 декабря 2014 года.
- ↑ Berry, M. J.; Banu, L.; Harney, J. W.; Larsen, P. R. Functional Characterization of the Eukaryotic SECIS Elements which Direct Selenocysteine Insertion at UGA Codons (англ.) // The EMBO Journal : journal. — 1993. — Vol. 12, no. 8. — P. 3315—3322. — PMID 8344267. — PMC 413599. Архивировано 20 сентября 2018 года.
- ↑ Kryukov, G. V.; Castellano, S.; Novoselov, S. V.; Lobanov, A. V.; Zehtab, O.; Guigó, R.; Gladyshev, V. N. Characterization of Mammalian Selenoproteomes (англ.) // Science. — 2003. — Vol. 300, no. 5624. — P. 1439—1443. — doi:10.1126/science.1083516. — PMID 12775843.
- ↑ Block, E. Garlic and Other Alliums: The Lore and the Science (англ.). — Royal Society of Chemistry, 2010. — ISBN 0-85404-190-7.
Литература[править | править код]
- Lobanov A. V., Hatfield D. L., Gladyshev V. N. Eukaryotic selenoproteins and selenoproteomes // Biochimica et Biophysica Acta. — 2009. — Vol. 1790, № 11. — P. 1424—1428. — doi:10.1016/j.bbagen.2009.05.014.
- Turanov A. A., Shpikalova M. A., Lobanov A. V., Fomenko D. E., Morrison H. G., Sogin M. L., Klobutcher L. A., Hatfield D. L., Gladyshev V. N. Genetic code supports targeted insertion of two amino acids by one codon (англ.) // Science. — 2009. — Vol. 323, no. 5911. — P. 259—261. — doi:10.1126/science.1164748.
- Zinoni F., Birkmann A., Leinfelder W., Bock A. Cotranslational insertion of selenocysteine into formate dehydrogenase from Escherichia coli directed by a UGA codon (англ.) // Proceedings of the National Academy of Sciences. — National Academy of Sciences, 1987. — Vol. 84, no. 10. — P. 3156—3160. — doi:10.1073/pnas.84.10.3156. — PMID 3033637. — PMC 304827.
Ссылки[править | править код]
- Одноклеточное поставило под сомнение одну из основных догм генетики. Лента.ру, 11 января 2009 г. (Дата обращения: 29 июля 2011)
- Пётр Баранов. Кодон в двух лицах. Газета.ру, 12 января 2009 г. (Дата обращения: 29 июля 2011)
- Александр Марков. Генетический код допускает разночтения. Элементы, 14 января 2009 г. (Дата обращения: 29 июля 2011)